
þ Sinomics 非编码RNA芯片
近年来大量研究表明非编码RNA在人类疾病调控中扮演重要角色。为开发疾病诊断标志物和筛选新药靶标带来新方向。同时,越来越多的新的转录组实验结果表明,基因组非编码序列有绝大部分是可以表达的。越来越多的事实证明转录出来的非编码RNA具有重要的生物学功能。
相关系列芯片服务:
Sinomics lncRNA芯片、Sinomics circRNA芯片、Sinomics ceRNA芯片
Sino Human lncRNA array V3.0
Sino human lncRNA array V3.0 是一款对转录产物进行检测的高通量芯片,检测范围包括lncRNA, mRNA。通过该芯片的检测,可以在全基因组范围内检测目前已知的差异RNAs分子,为后续的功能研究提供依据。
Sino Human lncRNA array V3.0芯片特点
² 设计平台:基于Agilent eArray平台设计,采用Agilent SurePrint喷墨打印技术合成,
² 稳定性好;
² 探针特点:经典 60 mer探针长度,检出率高,重复性好;
² 数据来源:以国际主流lncRNA数据库为基础,包括NCBI,Ensmble,NONCODE,lnciPedia,结合lncRNA疾病,lncRNA外泌体研究数据库注释,功能研究数据库整合,来源范围广,参考意义大;
² 检测范围:同时检测lncRNA和mRNA,并可实现两者数据关联。
Sino Human lncRNA array V3.0芯片参数
1. 芯片检测的RNA的数量分布
规格 | 探针长度 | ncRNA数目 | mRNA数目 |
4×180K | 60 nt | 91,614 | 25,353 |
2. 参考数据库来源及数量
数据库 | NCBI | Ensemble | Noncode V5 | Lncipedia5.0 |
检测数量 | 19,592 | 6,833 | 62,670 | 2,519 |
Sino Human lncRNA array V3.0芯片数据分析
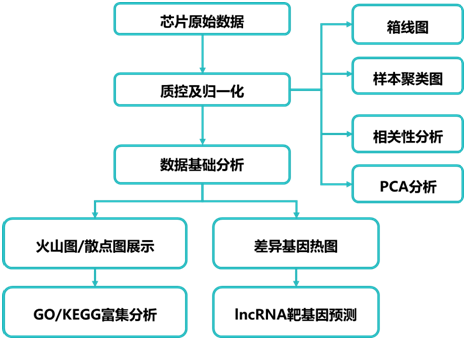
基础分析 | |
1. 原始数据处理分析 | 1)数据归一化 |
2)箱线图 | |
3)样本聚类图 | |
4)样本相关性分析 | |
5)主成份分析 (PCA) | |
2. 差异基因筛选 | 1)表达差异基因筛选 |
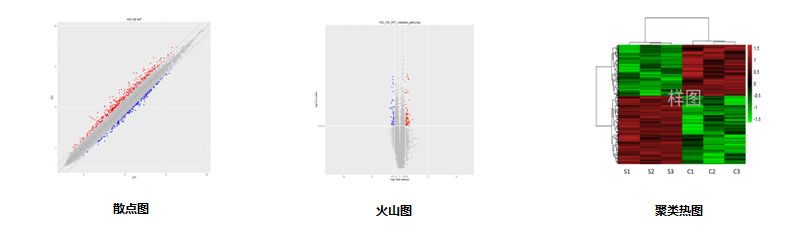
2)散点图 | |
3)火山图 | |
4)差异基因层级聚类热图 | |
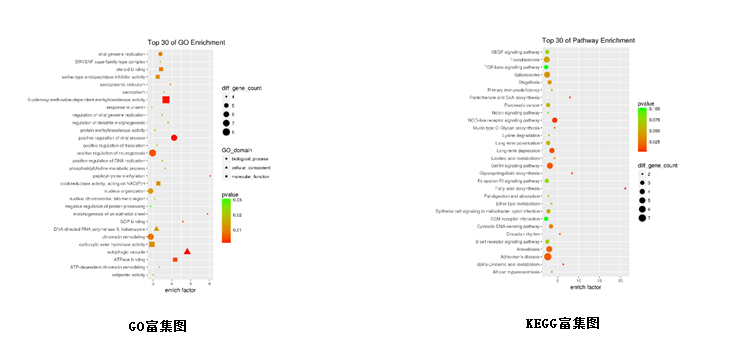
3. 差异mRNA功能分析(GO/KEGG) | |
4. lncRNA cis/trans靶基因预测 | |
5. 靶基因功能分析(GO/KEGG) | |
Sino Human lncRNA array V3.0芯片数据分析展示
1. 原始数据处理分析
2. 差异基因筛选
3. 差异mRNA功能分析(GO/KEGG)
4. lncRNA吸附miRNA预测
近年的研究表明,lncRNA分子富含microRNA结合位点,在细胞中起到miRNA海绵吸附(miRNA sponge)的作用,进而解除miRNA对其靶基因的抑制作用,升高靶基因的表达水平,这种机制称为内源竞争性RNA(ceRNA)机制。
我们通过对lncRNA与miRNA可能结合的位点分析,预测lncRNA吸附的miRNA分子,以便于从ceRNA角度研究lncRNA的分子功能。
5. lncRNA的cis/trans靶基因预测
Cis:选取与lncRNA距离小于10kb的gene作为cis作用的靶基因。
Trans:预测采用数据库是物种对应的gene序列数据库。先采用blast选择出序列上具有互补性或相似性的序列,再利用RNAplex计算两序列之间的互补能量,选择e≤−30的序列。
6. lncRNA疾病与外泌体研究注释
lncRNA与疾病的发生发展有着密切关系。虽然目前已知切确功能的lncRNA不多,但已有大量相关文献报道为研究lncRNA提供了参考。我们采用lncrnadisease(http://www.cuilab.cn/lncrnadisease)数据库对目前疾病中发现的lncRNA进行注释。
外泌体形成是由细胞膜内陷,形成内体(endosome),再形成多泡体(multivesicular bodies,MVB),最后分泌到胞外的囊泡结构。外泌体携带有母细胞的多种蛋白质、脂类、DNA和RNA(mRNA, lncRNAs, miRNAs, circRNA)等重要信息。我们采用exorbase (http://www.exorbase.org/)和exoCarta(http://www.exocarta.org/)数据库,对已有研究的外泌体中发现的lncRNA进行注释,为后续研究,特别是外泌体研究提供参考。
lncRNA常用数据库
² NCBI:https://www.ncbi.nlm.nih.gov/
² ENSEMNBL:http://asia.ensembl.org/index.html
² UCSC:http://genome.ucsc.edu/
² NONCODE:http://www.noncode.org/
² LNCipedia:https://lncipedia.org/
² LncRNADisease database: http://www.cuilab.cn/lncrnadisease
² CPC: http://cpc.cbi.pku.edu.cn/
中科普瑞科技服务团队参与lncRNA芯片发表相关文献
1 Pan Y, Chen J, Tao L. et al. Long noncoding RNA ROR regulates chemoresistance in docetaxel-resistant lung adenocarcinoma cells via epithelial mesenchymal transition pathway. Oncotarget. 2017, 8(20):33144-33158.
2 Wang W,, Gao F, Zhao Z, et al. Integrated analysis of lncRNAmRNA co-expression profiles in patients with moyamoya disease. Sci Rep. 2017, 7:42421.
Zhou M, Hou Y, Yang G,et al. LncRNA-Hh Strengthen Cancer Stem Cells Generation in Twist-Positive Breast Cancer via Activation of Hedgehog Signaling Pathway. Stem Cells. 2016 , 34(1):55-66.
3 Xu B, Shao Q, Xie K, et al. The Long Non-Coding RNA ENST00000537266 and ENST00000426615 Influence Papillary Thyroid Cancer Cell Proliferation and Motility. Cell Physiol Biochem. 2016 , 38(1):368-78.
4 Li Y, Wang X, Li M, et al. Long Non-Coding RNA Expression Profile in the Kidneys of Male, Low Birth Weight Rats Exposed to Maternal Protein Restriction at Postnatal Day 1 and Day 10. Plos One.2015,10(3):e0121587.
5 Li Y, Wang H, Sun T, et al. Biological characteristics of a new human glioma cell line transformed into A2B5+ stem cells. Mol Cancer. 2015,14(1):75.
6 Lan X, Zhang H, Wang Z, et al.Genome-wide analysis of long noncoding RNA expression profile in papillary thyroid carcinoma. Gene. 2015, 569(1):109-17.
7 Zeng QH, Wang Q, Chen X,et al. Analysis of lncRNAs expression in UVB-induced stress responses of melanocytes. J Dermatol Sci. 2016,81(1):53-60.
8 Li J, Li P, Zhao W, et al. Expression of long non-coding RNA DLX6-AS1 in lung adenocarcinoma. Cancer Cell Int. 2015, 15:48.
9 Xu WH, Zhang JB, Dang Z, et al. Long Non-coding RNA URHC Regulates Cell Proliferation and Apoptosis via ZAK through the ERK/MAPK Signaling Pathway in Hepatocellular Carcinoma. Int J Biol SCI. 2014, 10(7):664-76.
10 Hu P ,Yang JJ, Hou YX, et al. LncRNA expression signatures of twist-induced epithelial-to-mesenchymal transition in MCF10A cells. Cell Signal, 2014 (IF:4.471)
11 Wang Q, Fan H, Liu Y, et al. Curcumin enhances the radiosensitivity in nasopharyngeal carcinoma cells involving the reversal of differentially expressed long non-coding RNAs. Int J Oncol. 2014, 44(3):858-64.
12 Xing D, Liang JQ, Li Y, et al. Identification of long noncoding RNA associated with osteoarthritis in humans. Orthop Surg. 2014, 6(4):288-93.
13 Yang Q, Zhang S, Liu H, et al. Oncogenic role of long noncoding RNA AF118081 in anti-benzo[a]pyrene-trans-7,8-dihydrodiol-9,10-epoxide-transformed 16HBE cells. Toxicol Lett. 2014, 229(3):430-9.


