
一、Agilent Human miRNA芯片
安捷伦科技有限公司(Agilent Technologies Inc.)作为国际领先的生物芯片制造企业,利用其先进的芯片生产工艺,开发出具有独特设计的miRNA检测芯片,能特异性地检测出成熟的miRNA并能很好地区分高度同源的miRNA 分子。该平台还具有特异性高、灵敏度高等特点。可以广泛地应用于检测和比较各类样品的miRNA表达状态,并通过分析其调控的靶基因来深入分析其调控机制或生物学功能。
Agilent表达谱芯片特点
² 通过特殊的探针设计区分成熟miRNA和miRNA前体的芯片平台;
² 无需分离miRNA,直接用Total RNA 进行实验,只需100ng的Total RNA 即可用来进行标记实验(不包括质检所消耗的量),避免了分离、富集、放大等过程带来的影响;
² 样品适用范围广:组织、血清、血浆、FFPE、冰冻切片组织、脱落细胞等均可进行实验;
² 高特异性:特殊的专利探针设计能区别一个碱基的差异;
² 高灵敏度:检测丰度跨4个log,高可达6个log,只要存在6000个拷贝的分子就可以检出;
² 数据来源于miRBase 21.0版本,并方便及时更新;
² 人、大鼠、小鼠之外的动物物种,可以方便的通过earray进行个性化定制。
Agilent miRNA芯片类型及参数

Agilent表达谱芯片数据分析
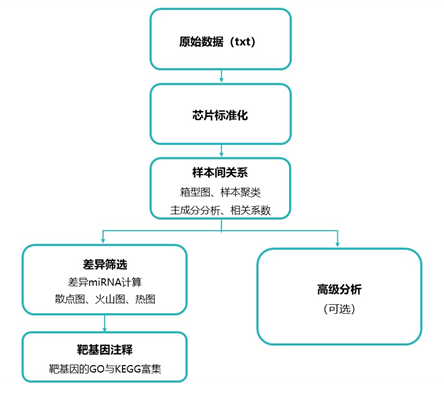
基础分析 | |
数据质控 | 1.各样本的箱线图 |
2.各样本的聚类图 | |
3.样本相关性图 | |
4. PCA图 | |
差异筛选 | 1.标准化后的microRNA总表 |
2.差异microRNA列表 | |
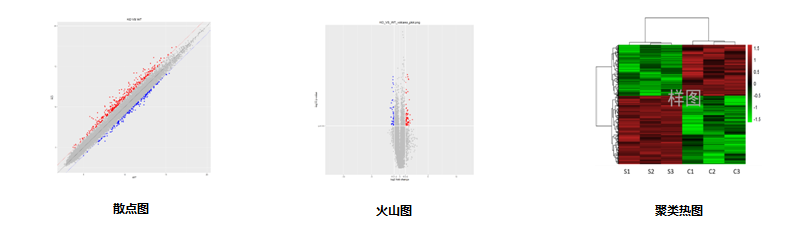
3.差异microRNA散点图 | |
4.差异microRNA火山图 | |
5.差异microRNA聚类热图 | |
功能注释 | 1.差异microRNA的靶基因注释 |
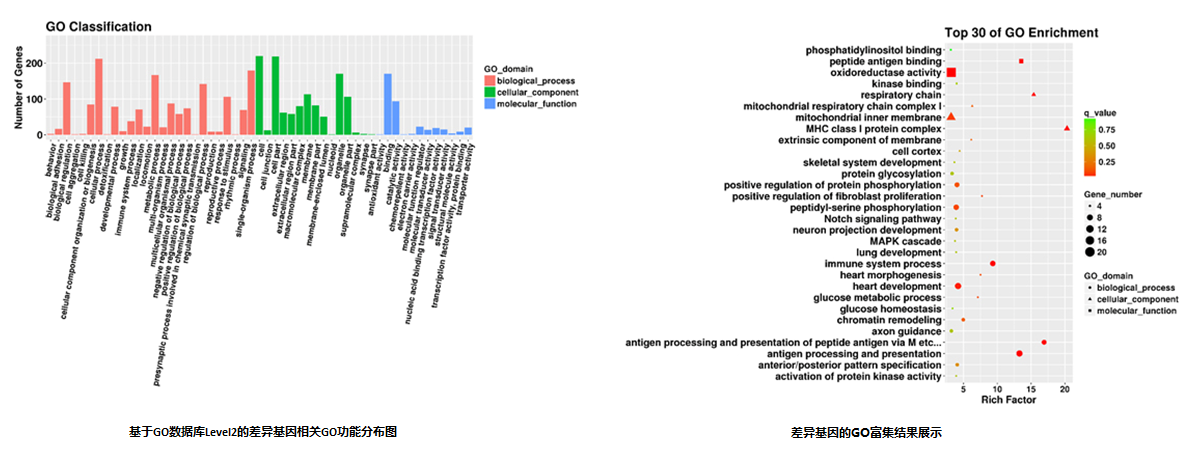
2. 靶基因的GO富集 | |
3.靶基因的KEGG富集 | |
高级分析 | |
1. 差异microRNA与lncRNA关系预测 | |
2. 差异microRNA与circRNA关系预测 | |
3. 差异microRNA与转录因子关系预测 | |
4.差异microRNA与靶基因的网络图 | |
5.差异microRNA与表达谱数据共表达计算 | |
6. 差异microRNA相关的ceRNA网络图 | |
Agilent表达谱芯片数据分析展示
1.原始数据处理分析
2. 差异基因筛选
3.靶基因功能分析(GO/KEGG)
4.miRNA靶基因预测
microRNA结合在靶基因的3’UTR,下调靶基因。miRNA靶基因预测有多种方法。我们主要利用TargetScan数据库关联microRNA的靶基因。
5.miRNA与靶基因网络图
将显著性功能与显著性Pathway所包含的靶基因取交集后与microRNA构建基因与microRNA调控网络(待确认),可以在全局的水平上直观的反应基因之间的相互关系,同时反映了基因调控网络的稳定性。根据网络中microRNA的位置函数计算出microRNA在网络中的关系强度,即microRNA的网络特征值。特征值高microRNA处于网络的枢纽性地位,该microRNA调控能力强,对网络结构和样本性状有重要的调控价值,同时从网络中也可以得到被microRNA调控的关键靶基因。
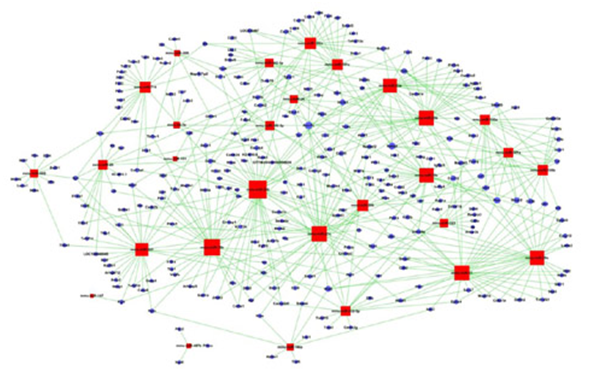
miRNA与靶基因网络图
6.miRNA-GO Network
miR-GO Network利用靶基因的功能注释与microRNA-mRNA靶向调控关系,构建microRNA功能调控的网络图。网络图可发现MicroRNA调控的多种基因功能,并通过网络分析,得到核心调控microRNA以及microRNA调控的核心基因功能。
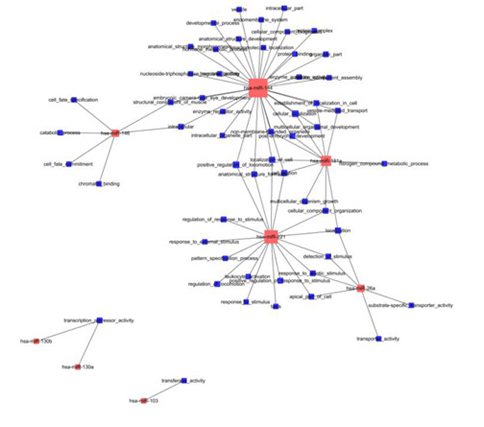
miRNA-GO Network
7.miRNA-Pathway Network
miR- Pathway Network与miR-GO Network类似.利用靶基因的Pathway间相互作用关系,与microRNA-mRNA靶向调控关系,构建miR- Pathway调控的网络图。网络图可发现MicroRNA调控的多种信号通路,并通过网络分析,得到核心调控microRNA以及microRNA调控的核心信号通路。
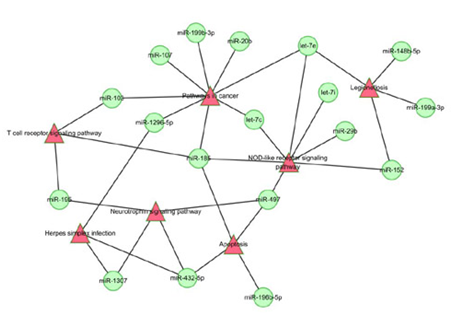
miRNA-Pathway Network
8.miRNA的转录因子的预测
为了研究miRNA的调控机制,首先预测miRNA的promoter区域,根据miRNA的promoter区域,利用HMM来预测结合的转录因子。
9.miRNA与表达谱芯片平台的联合分析
miRNA与表达谱芯片平台的联合分析除了和甲基化与表达谱芯片分析类似外,还可以将miRNA预测的靶基因和表达谱芯片的差异基因取交集,进行miRNA-DiffGene-Network。

miRNA-DiffGene-Network
二、中科普瑞基因芯片服务平台
高标准 — 拥有15年以上科技服务实验和分析技术经验的团队成员以GLP-like质量管理体系为您的基因芯片服务体系全线保驾护航。
高效率 — 样品质检合格后,15个工作日出具结果报告,专属生物信息和技术支持服务人员为您提供在线结果咨询和解释服务。
高质量 — 符合3大基因芯片厂商质量要求的服务质量体系,项目质量管理专员负责对您的项目全程跟踪和质控。

三、中科普瑞科技服务团队参与Agilent miRNA芯片发表相关文献
1. Ling Q, Xu X, Ye P, Xie H, Gao F, Hu Q, Liu Z, Wei X, Röder C, Trauzold A, Kalthoff H, Zheng S. The prognostic relevance of primary tumor location in patients undergoing resection for pancreatic ductal adenocarcinoma. Oncotarget(2017). IF5.008.
2. Jia HL, Liu CW, Zhang L, Xu WJ, Gao XJ, Bai J, Xu YF, Xu MG, Zhang G. Sets of serum exosomal microRNAs as candidate diagnostic biomarkers for Kawasaki disease. Sci Rep(2017). IF 5.228.
3. Wang Y, Liang J, Qin H, Ge Y, Du J, Lin J, Zhu X, Wang J, Xu J. Elevated expression of miR-142-3p is related to the pro-inflammatory function ofmonocyte-derived dendritic cells in SLE. Arthritis Res Ther(2016). IF3.979.
4. Shang FF, Xia QJ, Liu W, Xia L, Qian BJ, You L, He M, Yang JL, Wang TH. miR-434-3p and DNA hypomethylation co-regulate eIF5A1 to increase AChRs and to improve plasticity in SCT rat skeletal muscle. Sci Rep.(2016). IF 5.578.
5. Sun Y, Cai J, Yu S, Chen S, Li F . MiR-630 Inhibits Endothelial-Mesenchymal Transition by Targeting Slug in Traumatic Heterotopic Ossification. Sci Rep(2016). IF 5.578.
6. Dong R, Shen Z, Zheng C, Chen G, Zheng S. Serum microRNA microarray analysis identifies miR-4429 and miR-4689 are potential diagnostic biomarkers for biliary atresia. Sci Rep(2016). IF 5.578.
7. Zhao JL, Huang F, He F, Gao CC, Liang SQ, Ma PF, Dong GY, Han H, Qin HY.Forced Activation of Notch in Macrophages Represses Tumor Growth by Upregulating miR-125a and Disabling Tumor-Associated Macrophages.Cancer Res(2016). IF9.329.
8. Ye FG, Song CG, Cao ZG, Xia C, Chen DN, Chen L, Li S, Qiao F, Ling H, Yao L, Hu X, Shao ZM. Cytidine Deaminase Axis Modulated by miR-484 Differentially Regulates Cell Proliferation and Chemoresistance in Breast Cancer Cancer Res(2015). IF9.284.
9. Yang B, Huang J, Liu H, Guo W, Li G. miR-335 directly, while miR-34a indirectly modulate survivin expression and regulate growth, apoptosis, and invasion of gastric cancer cells. Tumour Biol(2015). IF 3.611.
10. Wen J, Luo K, Liu H, Liu S, Lin G, Hu Y, Zhang X, Wang G, Chen Y, Chen Z, Li Y, Lin T, Xie X, Liu M, Wang H, Yang H, Fu J. MiRNA Expression Analysis of Pretreatment Biopsies Predicts the Pathological Response of Esophageal Squamous Cell Carcinomas to Neoadjuvant Chemoradiotherapy.Ann Surg(2015). IF 8.327.
11. Zou Z, Chen J, Liu A, Zhou X, Song Q, Jia C, Chen Z, Lin J, Yang C, Li M, Jiang Y, Bai X. mTORC2 promotes cell survival through c-Myc-dependent up-regulation of E2F1. J Cell Biol(2015). IF9.834.
12. Yanmin X, Ji H, Leina M, Juanjuan S, Junjie S, Zhi Y, Limei L, Yongli L, Chao Y, Cheng Q.MicroRNA-122 confers sorafenib resistance to hepatocellular carcinoma cells by targeting IGF-1r to regulate RAS/RAF/ERK signaling pathways.Cancer Lett(2015). IF5.621.
13. Wang X, Chen X, Han W, Ruan A, Chen L, Wang R, Xu Z, Xiao P, Lu X, Zhao Y, Zhou J, Chen S, Du Q, Yang H, Zhang X. miR-200c Targets CDK2 and Suppresses Tumorigenesis in Renal Cell Carcinoma.Mol Cancer Res(2015). IF4.38.


